Dam-Methylase
Die Dam-Methylase (Abkürzung für Desoxyadenosin-Methylase) ist ein Enzym, das eine Methylgruppe an den Adenin-Rest der GATC-Sequenz in bakterieller DNA koppelt.[1][2] Der Tochterstrang wird nicht sofort methyliert, was die Zelle für Reparaturen nutzen kann.[3]
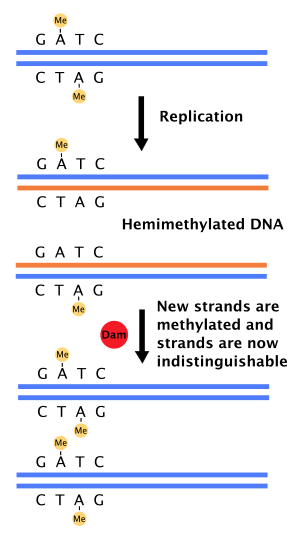
Rolle bei der Reparatur von Basen-Fehlpaarungen
BearbeitenTritt bei der DNA-Synthese ein Fehler bei der Basenpaarung auf, oder wird eine zusätzliche Base eingebaut, leitet die molekulare Zellmaschinerie die Korrektur durch DNA-Mismatch-Reparaturproteine ein. Dafür ist es notwendig, den Vorlage- vom neusynthetisierten Tochterstrang unterscheiden zu können. In Bakterien werden die DNA-Stränge von der Dam-Methylase methyliert. Nach Neusynthese besitzt zunächst nur die Vorlage eine Methylgruppe; daher spricht man vom Zustand der „Hemimethylierung“. Ein Reparaturenzym bindet an fehlgepaarte Basen und rekrutiert den Faktor MutL, der anschließend die MutH-Endonuklease aktiviert. MutH bindet hemimethylierte DNA spezifisch an der Sequenz GATC und schneidet den Tochterstrang hier. Dadurch können das Entwindungsenzym Helikase und Exonukleasen die fehlerhaften Stellen im neusynthetisierten Strang entfernen.[3][4] Die Neusynthese des entfernten Strangabschnittes erfolgt durch DNA-Polymerase III.
Rolle bei der Regulation der Replikation
BearbeitenDie Hybridisierung der DNA-Polymerase mit dem Replikationsstartpunkt (oriC) in Bakterien wird streng kontrolliert, um sicherzustellen, dass die DNA-Synthese nur einmal pro Zellzyklus stattfindet (ansonsten hätte die Zelle ein Vielfaches der üblichen genetischen Information). Die Kontrolle findet unter anderem durch die langsame Hydrolyse von ATP durch DnaA statt. DnaA ist ein Protein, das an Sequenzwiederholungen im oriC bindet, um die Replikation zu starten. Die Dam-Methylase spielt ebenfalls eine Rolle, da oriC in Escherichia coli elf GATC-Sequenzen besitzt. Kurz nach dem Abschluss der DNA-Synthese liegt oriC hemimethyliert vor und wird von dem Protein SeqA gebunden[5]. Die Bindung von SeqA ist spezifisch für hemimethylierte GATCs. SeqA blockiert auf diese Weise den oriC und verhindert eine Bindung von DnaA. Nach einiger Zeit löst sich SeqA vom oriC, die GATCs können re-methyliert werden und DnaA wieder binden.
Rolle bei der Regulation der Genexpression
BearbeitenDie Dam-Methylase spielt auch eine Rolle bei der Aktivierung und Repression der Transkription. In E. coli sind GATC-Sequenzen stromabwärts methyliert und fördern so die Transkription. Beispielsweise wurde beschrieben, dass in uropathogenen Coli-Bakterien die Methylierung von zwei GATC-Stellen neben dem PAP-Promotor reguliert wird.[6]
Siehe auch
BearbeitenEinzelnachweise
Bearbeiten- ↑ Marinus MG, Morris NR: Isolation of deoxyribonucleic acid methylase mutants of Escherichia coli K-12. In: J. Bacteriol. 114. Jahrgang, Nr. 3, Juni 1973, S. 1143–50, PMID 4576399, PMC 285375 (freier Volltext).
- ↑ Geier GE, Modrich P: Recognition sequence of the dam methylase of Escherichia coli K12 and mode of cleavage of Dpn I endonuclease. In: J. Biol. Chem. 254. Jahrgang, Nr. 4, Februar 1979, S. 1408–13, PMID 368070.
- ↑ a b Barras F, Marinus MG: The great GATC: DNA methylation in E. coli. In: Trends in Genetics. 5. Jahrgang, 1989, S. 139–143, doi:10.1016/0168-9525(89)90054-1.
- ↑ Løbner-Olesen A, Skovgaard O, Marinus, MG: Dam methylation: coordinating cellular processes. In: Curr. Opin. Microbiol. 8. Jahrgang, Nr. 2, April 2005, S. 154–160, doi:10.1016/j.mib.2005.02.009, PMID 15802246.
- ↑ Waldminghaus T and Skarstad K: The Escherichia coli SeqA protein. Plasmid. 2009 May, 61(3):141-50. doi:10.1016/j.plasmid.2009.02.004
- ↑ Casadesús, J und Low, D: Epigenetic Gene Regulation in the Bacterial World. Microbiol Mol Biol Rev. 2006 Sep; 70(3): 830–856. doi:10.1128/MMBR.00016-06